现代生物技术,尤其是合成生物学的快速发展,使得我们能够通过基因改造赋予植物新的性状。植物基因组的精确修饰可以通过同源重组或基因编辑实现,而新基因也可以通过随机整合的方式导入植物基因组。然而,在高等植物中,通过同源重组进行基因靶向(gene targeting)修饰的效率通常极低,这成为精确修饰植物基因组的主要障碍之一。农杆菌介导的T-DNA随机整合到植物基因组是一种常用的基因导入工具,但筛选转基因植物需要使用选择标记(selection marker),而公众对选择标记可能对人类健康造成的潜在风险表示担忧。目前,欧盟超过一半的国家已正式禁止转基因(genetically modified)作物的种植。因此,开发无选择标记的DNA导入技术对于安全转基因应用具有重要意义,相关研究也备受关注。尽管已有多种策略被探索,如共转化、转座元件和位点特异性重组等,但这些方法通常操作繁琐且效率不高。

2025年2月5日,Plant Direct在线发表上海师范大学王水团队题为“Delivery of Marker-FreeDNA to Plant Genome by the Transgenic Selection-Associated Fragment Elimination(T-SAFE) System”的研究论文。该研究成功建立了一种简单、高效且安全的方法,能够向植物基因组递送大片段DNA(>10 kb),为植物基因改造提供了新的技术手段。
该研究开发了一种基于CRISPR/Cas9技术的简化高效系统,称为Transgenic Selection-Associated Fragment Elimination (T-SAFE)系统(图1)。T-SAFE系统由四个模块组成:选择标记、CRISPR/Cas9、间隔序列加原间隔序列相邻基序(SP)和递送的DNA。其中,选择标记和CRISPR/Cas9模块合称为SCC。SCC两侧是两个源自果蝇Ebony基因的相同SP(SP-F和SP-R,序列方向相反),这些序列能够在外源DNA整合到植物基因组时,有效引导Cas9切割包含选择标记基因的SCC,从而实现选择标记的自我消除。此外,为了抑制细菌中功能性Cas9蛋白的产生,研究团队将马铃薯ST-LS1基因的IV2内含子整合到Cas9基因中;同时,为了避免在非生殖植物细胞中发生SCC切割,Cas9基因由生殖细胞特异性启动子或诱导型启动子驱动。这些创新设计使T-SAFE系统在拟南芥转基因植物中选择标记的消除效率达到10-30%,在水稻中达到5-8%,且DNA递送能力超过10 kb。这一方法为植物基因改造提供了一种安全可靠的新途径。
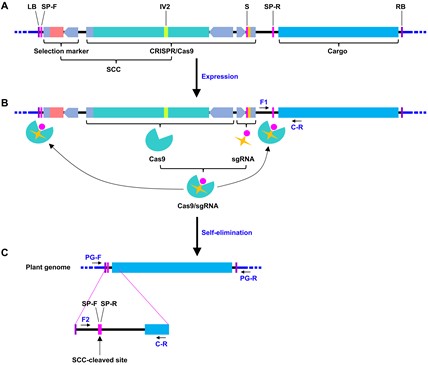
图1. T-SAFE系统示意图。
(A)T-SAFE系统由四个模块组成:选择标记(selection marker)、CRISPR/Cas9、SP(SP-F和SP-R,序列方向相反)和递送的DNA(Cargo)。(B)DNA整合到植物基因组后,sgRNA引导Cas9有效切割SCC两端的SP。(C)自我消除SCC后,无选择标记的目标DNA成功地送到植物基因组。引物对F1/C-R和F2/C-R分别用于检测植物中整合片段是否含有SCC。采用TAIL-PCR扩增整合片段侧翼的植物基因组序列。根据TAIL-PCR结果,设计了两对引物PG-F/C-R和PG-F/PG-R,用于鉴定基因组DNA片段与SCC切割后的目标DNA片段的整合情况。
上海师范大学生命科学学院卓越创新班本科生杨奕为论文第一作者,王水研究员为通讯作者。实验室研究生常欢、潘镭文、郭东北、彭顺、毛婷和张悦慧等同学参与了本项研究。相关工作得到了国家自然科学基金委和上海市植物种质资源开发协同创新中心等项目的资助。
供 稿:王 水
责任编辑:陈婉娴


